[ad_1]

Los genes saltarines, como el que da a este maíz su color distintivo, han proporcionado un nuevo mecanismo para la edición de genes.Crédito: Getty
Puede haber una peculiaridad molecular presente en las bacterias. Tiene la clave para rediseñar los genomas a voluntad.Esto permite a los investigadores insertar, eliminar o invertir grandes secciones de ADN.
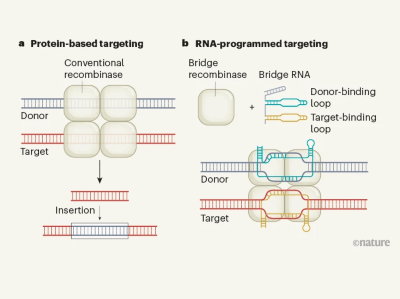
La técnica se describe en tres artículos de investigación publicados este mes en naturaleza1,2 Y Comunicaciones de la naturaleza3explota la capacidad natural de secuencias genéticas móviles, llamadas genes saltadores, Insertarse en genomas..
Guiado por una molécula de ARN llamada “puente” de ARN o “ARN de búsqueda”, se ha demostrado que el sistema puede editar genes en bacterias y en reacciones de probeta, pero aún no está claro si se puede adaptar para funcionar en humanos. células. . Si es posible, podría ser revolucionario, dado su pequeño tamaño y su capacidad para realizar cambios genéticos de miles de bases de longitud (mucho más grandes de lo que es práctico con el sistema de edición del genoma CRISPR-Cas9) sin romper el ADN.
“Si esto funciona en otras células, cambiará las reglas del juego”, dice Sandro Fernández-Ataide, biólogo estructural de la Universidad de Sydney en Australia y autor del estudio. Comunicaciones de la naturaleza “Abre un nuevo campo en la edición de genes”.
Tesoros transportables
Como muchas celebridades, el ascenso de CRISPR-Cas9 a la fama ha estado plagado de titulares engañosos. Aunque este método puede utilizarse para reescribir pequeñas partes del genoma, no es el sistema versátil de cortar y pegar que algunas noticias han presentado. Esta técnica se utiliza a menudo para cambiar una o varias bases del ADN; esto normalmente requiere romper el ADN primero y luego confiar en los sistemas innatos de reparación del ADN de la célula para generar el cambio deseado. Sin embargo, esto abre la puerta a daños genéticos colaterales no deseados a medida que la célula lleva a cabo su reparación.
A medida que CRISPR avanza hacia la medicina humanaLos investigadores buscan ampliar su conjunto de herramientas de edición del genoma para poder insertar genes completos o incluso múltiples genes en la ubicación de su elección. Hacerlo les permitirá desarrollar un tratamiento que trate a personas que tienen múltiples mutaciones en un solo gen, en lugar de atacar cada mutación con un enfoque personalizado. La capacidad de modificar muchos genes puede permitir a los investigadores hacer esto Ingeniería de células inmunes para atacar el cáncer De múltiples maneras, todo ello manteniendo el control sobre dónde se insertan estos genes en el genoma.
“Lo que realmente queremos hacer en el futuro es poder diseñar secciones enteras de nuestro genoma, no bases individuales”, dice Patrick Hsu, bioingeniero del Ark Institute, una organización sin fines de lucro en Palo Alto, California, y autor de ambos libros. naturaleza Hojas.
Para buscar herramientas, Hsu y sus colegas examinaron una clase diversa de enzimas que permiten que los elementos móviles del ADN de las bacterias salten de un lugar a otro. Se decidieron por una familia de artículos transportables llamada IS110.
El equipo descubrió que las enzimas de la familia IS110 utilizan un sistema de direccionamiento complejo e inusual basado en ARN. Un extremo del ARN se une a un trozo de ADN que se insertará en el genoma, y el otro extremo se une a un trozo de ADN en el lugar del genoma donde irá la carga. Debido a que el ARN une las dos partes del ADN, el equipo llamó a estas moléculas “puente de ARN”.
Al cambiar las secuencias en cada extremo de este puente, los investigadores pudieron programar las enzimas IS110 para insertar la carga útil de su elección donde querían en el genoma. Utilizaron el sistema para insertar con precisión un trozo de ADN de unas 5.000 bases de largo en el genoma bacteriano. Escherichia coliy para escindir y revertir otro trozo de ADN de bacterias coli Genoma.
Trabajando independientemente de Hsu, Atayde y sus colegas han caracterizado la bioquímica de las moléculas IS110, así como de moléculas de otra familia llamada IS1111, que utilizan un mecanismo similar y también pueden programarse. A estos mediadores de ARN los llaman “ARN de búsqueda”.
Identificar y explotar estos mecanismos es un logro notable, dice Elizabeth Kellogg, que estudia elementos móviles del ADN llamados transposones en el Hospital de Investigación Infantil St. Jude en Memphis, Tennessee. “A todo el mundo le encanta la capacidad de los transposones para insertar grandes cargas de ADN, pero hacerlos programables y específicos para un sitio es muy difícil”, añade.
Señala que otros sistemas de transporte que los investigadores han explorado para la edición del genoma son más complejos y, a menudo, constan de múltiples proteínas. En otro artículo de investigación publicado en naturaleza Este mes, los investigadores determinaron cómo los componentes clave de algunas de estas complejas máquinas forman una estructura compleja conocida como transposoma, que funciona con una enzima llamada transposasa para permitir que los elementos genéticos móviles se muevan por el genoma.4.
La medición importa
Del mismo modo, los esfuerzos por diseñar sistemas basados en CRISPR para realizar grandes manipulaciones en el genoma a menudo también requieren múltiples proteínas o la fusión de la enzima Cas con otra proteína. Por ejemplo, un artículo publicado el 26 de junio en celúla Describe un método para replicar partes del genoma de hasta 100 millones de bases (más grandes que algunos cromosomas humanos) utilizando una proteína Cas9 ligada a una enzima que puede copiar la secuencia del donante.5.
Enzimas programables guiadas por ARN para la edición del genoma de próxima generación
Por el contrario, los sistemas IS110 e IS1111 requieren solo una proteína, es decir Menos de la mitad del tamaño de muchas enzimas Cass. Utilizado en sistemas de edición del genoma CRISPR. Esta diferencia de tamaño es importante para las aplicaciones médicas: los virus que a menudo se utilizan para introducir componentes de edición del genoma en células humanas tienen una capacidad de carga limitada.
Pero los sistemas CRISPR también tienen la ventaja de la versatilidad, afirma Zhengzhou Long, bioingeniero de NYU Langone Health en la ciudad de Nueva York. Algunas enzimas Cas funcionan en casi todos los tipos de células que se han estudiado.
Long dice que es “hermoso” trabajar con el IS110 y el IS1111. “Pero realmente espero que dentro de unos meses digan que este tratamiento es eficaz en ratones”, añade. “Entonces tomemos una taza de café”.
Hasta ahora, ISIS110 Los miembros de esta familia no parecen funcionar bien en células de mamíferos, dice Hiroshi Nishimasu, biólogo estructural de la Universidad de Tokio que trabajó con Hsu para determinar el mecanismo por el cual la enzima IS110 se dirige al ADN. El equipo ahora está intentando diseñar esta enzima para que funcione mejor en células de mamíferos. Independientemente de su éxito allí, el mecanismo IS110 se destaca como una forma nueva y “elegante” mediante la cual los elementos móviles del ADN pueden moverse por el genoma, dice Nancy Craig, vicepresidenta senior de SalioGen Therapeutics, una empresa de biotecnología en Lexington, Massachusetts. tiene como objetivo desarrollar herramientas de edición del genoma utilizando elementos transponibles de mamíferos.
“La Madre Naturaleza ha encontrado muchas soluciones a este problema”, afirma. “Hemos encontrado algunas soluciones, pero hay muchas más por delante”.
[ad_2]
Source Article Link