[ad_1]

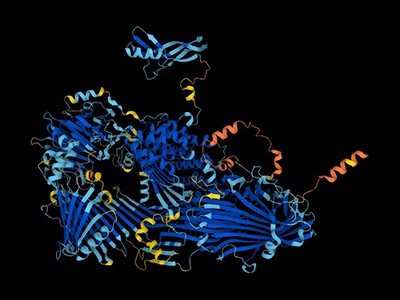
David Becker, Demis Hassabis y John Gumper (de izquierda a derecha) ganaron el Premio Nobel de Química por desarrollar herramientas computacionales que pueden predecir y diseñar estructuras de proteínas.Crédito: Fundación BBVA
Por primera vez, y quizás no la última, un avance científico posible gracias a la inteligencia artificial ha sido reconocido con el Premio Nobel. El Premio Nobel de Química 2024 fue otorgado a John Gamper y Demis Hassabis de Google DeepMind en Londres, por desarrollar un sistema revolucionario. Una herramienta de inteligencia artificial para predecir estructuras de proteínas se llama AlphaFoldy David Becker, de la Universidad de Washington en Seattle, por su trabajo en diseño computacional de proteínas, que se completó Al lo ha reforzado en los últimos años.
“Espero que cuando miremos a AlphaFold, sea la primera prueba del asombroso potencial de la inteligencia artificial para acelerar los descubrimientos científicos”, dijo Hassabis en una conferencia de prensa en DeepMind el 9 de octubre. “Es irreal en este momento”.
¿Qué sigue para AlphaFold y la revolución del plegado de proteínas con IA?
El efecto AlphaFold que fue Fue revelado hace sólo unos años.Fue nada menos que transformador. Esta herramienta ha puesto a disposición de los investigadores estructuras de proteínas (a menudo, pero no siempre, extremadamente precisas) con solo tocar un botón, y ha permitido experimentos que habrían sido inimaginables hace una década. “Es una gran revolución”, dice Christine Orengo, bióloga computacional del University College de Londres, cuyo laboratorio ha utilizado estructuras predichas por AlphaFold para revelar nuevas proteínas.
“Ha sido un sueño desde hace mucho tiempo que aprendamos a predecir la estructura tridimensional de las proteínas conociendo sus secuencias de aminoácidos”, dijo Heiner Linke, jefe del Comité Nobel, que investiga la nanociencia en la Universidad de Lund en Suecia. Durante varias décadas, esto se consideró imposible”. Durante el anuncio del premio. Añadió que los ganadores de este año “lograron descifrar el código”. Los tres ganadores comparten un premio acumulado de 11 millones de coronas suecas (1 millón de dólares).
Inteligencia artificial premiada
DeepMind lanzó AlphaFold por primera vez en 2018, cuando ganó un concurso de predicción de la estructura de proteínas llamado Evaluación crítica de la predicción de la estructura de proteínas (CASP). Pero fue la segunda iteración de la red neuronal de aprendizaje profundo, Fue presentado a finales de 2020.Esto realmente ha revolucionado las ciencias de la vida. Muchas de las predicciones de AlphaFold2 en CASP fueron tan precisas que no se podían distinguir de las estructuras proteicas resueltas experimentalmente.
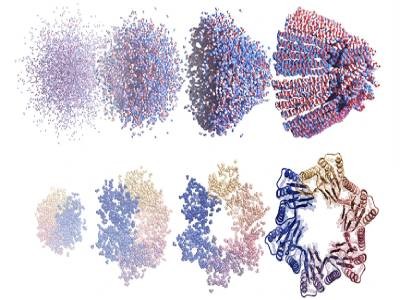
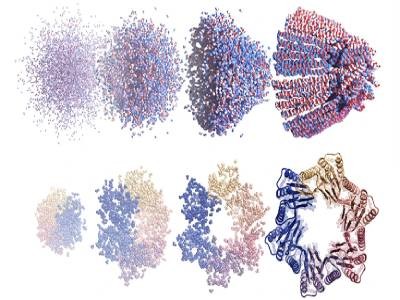
Hassabis, cofundador y director ejecutivo de DeepMind, y Jumper, jefe del equipo AlphaFold, lideraron el desarrollo de AlphaFold2. Para predecir las estructuras de las proteínas, una red neuronal combina datos de bibliotecas de cientos de miles de estructuras y millones de secuencias de proteínas relacionadas, que contienen información sobre sus formas.
Cómo ganar un Premio Nobel: ¿Qué tipo de científicos ganan medallas?
En particular, el éxito de AlphaFold se debe en gran parte al Protein Data Bank, un depósito de acceso gratuito de más de 200.000 estructuras de proteínas determinadas mediante métodos que incluyen cristalografía de rayos X y microscopía crioelectrónica. “Es una lección de humildad cada vez que entrenamos [AlphaFold] A lo largo de años de esfuerzo. “Cada punto de datos son años de esfuerzo por parte de alguien”, dijo Jumper en la conferencia de prensa de DeepMind.
En 2021, Mente profunda Hizo que el código base para AlphaFold2 esté disponible gratuitamenteAdemás de los datos necesarios para entrenar el modelo. eso Base de datos AlphaFoldcreado en colaboración con el Instituto Europeo de Bioinformática del Laboratorio Europeo de Biología Molecular en Hinxton, Reino Unido, ahora Contiene las estructuras de casi todas las proteínas de todos los organismos representados en bases de datos genéticas.alrededor de 214 millones de predicciones en total. Este año, la compañía dio a conocer La tercera versión de AlphaFoldque puede diseñar otras moléculas que interactúen con proteínas como medicamentos.
La revolución lanzada por Jumper, Hassabis y sus colegas aún está en sus inicios, y es posible que no se conozca el impacto total de AlphaFold en la ciencia hasta dentro de años. De hecho, esta herramienta ayuda a los científicos a obtener nuevos conocimientos.
Un equipo pionero utilizó esta herramienta, combinada con datos experimentales, para dibujar un mapa. Complejo de poros nuclearesuna de las máquinas más grandes de nuestras células que mueve moléculas dentro y fuera del núcleo. El año pasado, dos equipos exploraron toda la base de datos AlphaFold para descubrir los rincones más oscuros del mundo de las proteínas, identificando nuevas familias de proteínas y pliegues y conexiones sorprendentes en la maquinaria de la vida.
“Todo el mundo de las proteínas”: la IA predice la forma de casi todas las proteínas conocidas
Muchos investigadores esperan que AlphaFold y otras herramientas de inteligencia artificial que inspiró transformen la medicina, pero ese ya es el caso. no esta claro aun Cómo, o si, AlphaFold simplificará el costoso proceso de varios pasos para desarrollar medicamentos seguros. Los científicos que sientan las bases para nuevas vacunas encuentran que AlphaFold es increíblemente útil Y en algunos casos, un cambio de juego. Pero AlphaFold complementa los estudios experimentales y otros enfoques para mapear y modificar la estructura de proteínas virales para su uso en vacunas.
Para la mayoría de los investigadores, la estructura predicha es el comienzo de un estudio, no el final, dice Jan Kosinski, modelador estructural del Laboratorio Europeo de Biología Molecular (EMBL) en Hamburgo, Alemania. “Al principio se temía que sustituyera a la biología estructural, que la gente perdiera su empleo, etc. De hecho, ocurrió todo lo contrario”, añade.
Uno de los mayores impactos de la herramienta ha sido un cambio en la mentalidad de los biólogos, “que dicen que las computadoras son cosas que pueden producir hipótesis útiles”, dice David Jones, bioinformático de la University College London que colaboró con DeepMind en la primera versión de AlphaFold a partir de en 2016. Que se puede probar en el laboratorio.
Creando nuevas proteínas
Más de dos décadas antes de que DeepMind comenzara a trabajar en AlphaFold, el biofísico computacional David Becker y sus colegas desarrollaron una herramienta de software llamada Rosetta que modelaba estructuras de proteínas utilizando principios físicos. La herramienta compara pequeñas partes de múltiples estructuras y secuencias de proteínas existentes para determinar qué secuencias de proteínas pueden plegarse en una conformación específica.
Inicialmente, Rosetta se aplicó para predecir estructuras de proteínas y estuvo entre las principales entradas en muchos CASP, antes de que AlphaFold dominara. Pero Becker pronto se dio cuenta de que se podía invertir el paradigma para diseñar proteínas completamente nuevas.
Las herramientas de IA están diseñando proteínas completamente nuevas que podrían transformar la medicina
La herramienta tuvo un éxito temprano Diseñar nuevas proteínasincluidos nuevos tipos de enzimas, proteínas que pueden unirse estrechamente a otras moléculas y nanopartículas de proteínas autoensambladas que se asemejan a los virus (una de las cuales sirvió de base para una vacuna COVID-19 aprobada).
Cuando se anunció AlphaFold2, pero aún no se lanzó, Baker y su equipo, incluido el químico computacional Minkyung Baek, ahora en la Universidad Nacional de Seúl en Corea del Sur, se propusieron comprender el programa y aplicar algunos de sus trucos a una versión anterior basada en IA. del software. Rashid. El rendimiento de la primera versión de la red RoseTTAFold resultante fue casi idéntico al de AlphaFold2. Desde 2021, sus desarrolladores y otros científicos han mejorado continuamente ambas redes para afrontar nuevos desafíos, como predecir la estructura de complejos de múltiples y diferentes proteínas que interactúan.
En los últimos años, el equipo de Becker ha sido particularmente prolífico en la aplicación del aprendizaje automático a la razón de ser de su laboratorio: Creando nuevas proteínas nunca antes vistas en la naturaleza. La herramienta desarrollada recientemente por el equipo de Baker, que combina RoseTTAFold con redes neuronales omnipresentes generadoras de imágenes, ha supuesto un cambio radical en la capacidad de los investigadores para diseñar proteínas.
Avance rápido
Estas herramientas fueron un tremendo acelerador y democratizador, dice Sergei Ovchinnikov, biólogo evolutivo del Instituto Tecnológico de Massachusetts en Cambridge, que hizo su doctorado en el laboratorio de Becker. Rosetta solía tardar semanas en ejecutarse en cientos de procesadores para llegar a un diseño de proteína, una tarea que las herramientas más nuevas basadas en inteligencia artificial pueden realizar en segundos. “Ahora todo el mundo puede diseñar una proteína”, afirma.
“Me siento profundamente inspirado por otros en este campo y por la gente con la que he trabajado”, dijo Becker, hablando por teléfono durante el anuncio del Premio Nobel. “Me he subido a hombros de gigantes”.
Premio Nobel de Medicina por “microARN” que regula genes
Martin Steiniger, biólogo computacional de la Universidad Nacional de Seúl en Corea del Sur, compara el impacto de AlphaFold, RoseTTAFold y otras herramientas de inteligencia artificial biológica con el de las misiones Apolo a la Luna, al mostrar lo que la ingeniería puede lograr. “Es un momento similar para la predicción de estructuras y el campo de la biología estructural, donde simplemente se ve lo que es posible”, dice.
Pocos se sorprendieron por la decisión del Comité Nobel. En cuanto a Baker, Jones dice: “La mayoría de la gente pensó que tenía que ver con la cantidad de trabajo que había realizado en esta área”. Gamper, que sabía que él y Hassabis estaban en las listas de muchas personas, dijo en la rueda de prensa que no había podido dormir la noche anterior al anuncio de hoy.
Para Jumper, las estructuras predecibles que ofrece AlphaFold crean nuevas oportunidades para el descubrimiento científico. Millones de científicos ya han utilizado estas herramientas y espera que no pase mucho tiempo antes de que alguien reciba una llamada de Suecia. “El momento en que sentiré la misma emoción será el Premio Nobel por el trabajo realizado con AlphaFold”, dijo.
[ad_2]
Source Article Link