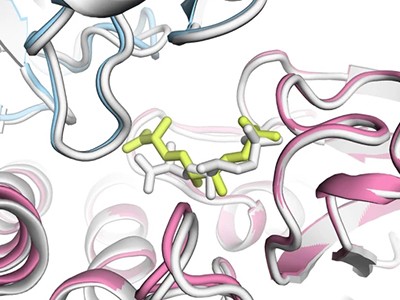
Un sábado por la mañana, a mediados de agosto, Alex Naka se embarcó en lo que describió como un “mini-hackathon” en la cocina de su novia. Impulsado por su computadora portátil, un poco de café y, en un momento dado, alrededor de 80 procesadores de inteligencia artificial (IA) basados en la nube, creó Docenas de proteínas diseñadas por computadora Diseñado para bloquear un receptor celular que está mutado en algunos tumores.
Naka, que trabaja entre semana como ingeniero de proteínas en una empresa de tecnología médica en Alameda, California, inscribió sus 10 creaciones prometedoras en un concurso de diseño de proteínas recientemente lanzado y las vio ascender a la cima de la clasificación.

El premio de química es para los desarrolladores de AlphaFold AI, que predice estructuras de proteínas
El concurso, organizado por una startup de biotecnología llamada Adaptyv Bio en Lausana, Suiza, es uno de al menos cinco que han surgido durante el último año. La mayoría de las personas que participan en competiciones hacen ejercicio. Herramientas de inteligencia artificial como AlphaFold Y los “modelos de lenguaje proteico” inspirados en chatbots han ganado popularidad y poder. Tres de los investigadores detrás de algunas de estas herramientas fueron homenajeados El Premio Nobel de Química de este año Por sus esfuerzos. Estos elogios provienen, en parte, de la esperanza de que las proteínas recién creadas puedan servir como fármacos, enzimas industriales o reactivos de laboratorio más eficaces.
Pero la mutación en las proteínas de diseño ha generado mayormente confusión, dicen los científicos. La gente los produce más rápido de lo que pueden fabricarse y probarse en laboratorios, lo que dificulta saber qué métodos son realmente efectivos.
Las competiciones han impulsado importantes avances científicos en el pasado, especialmente en el campo de la predicción de la estructura de las proteínas. Este último conjunto de concursos atrae a personas de todo el mundo a campos relacionados. El campo del diseño de proteínas. Bajando la barrera de entrada. También puede acelerar el ritmo de validación y desarrollo de estándares y potencialmente ayudar a fortalecer la comunidad. “Esto impulsará el campo hacia adelante y probará los métodos más rápidamente”, afirma Noelia Firouz Kababi, bióloga computacional del Centro de Regulación Genómica de Barcelona, España.
Pero los científicos dicen que las competiciones deben superar algunos obstáculos, como identificar qué problemas abordar y determinar cómo seleccionar objetivamente a los ganadores. Es importante acertar con la fórmula. “Estas competiciones pueden dañar” un campo si no se implementan adecuadamente, afirma Burkhard Rust, biólogo computacional de la Universidad Técnica de Munich en Alemania.
Competitivo por diseño
Los concursos de diseño de proteínas están inspirados en parte en un concurso de hace 30 años que ayudó a lanzar la revolución en la inteligencia artificial biológica. Desde 1994, la Evaluación Crítica de Predicción de Estructuras (CASP) ha desafiado a los científicos a predecir la forma tridimensional de las proteínas a partir de sus secuencias de aminoácidos. Los ganadores del concurso, fundado por el biólogo computacional John Molt de la Universidad de Maryland en Rockville, y Krzysztof Fidelis, biólogo computacional de la Universidad de California, Davis, se determinan comparando predicciones computacionales con modelos estructurales inéditos.
En 2018, DeepMind, con sede en Londres (ahora Google DeepMind), ganó CASP con su primera versión de una herramienta de predicción de la estructura de proteínas. Pliegue alfa. La próxima iteración, AlphaFold 2, Tuvo un buen desempeño en 2020. Mollet declaró que el problema de predecir estructuras proteicas simples se había resuelto en gran medida. Desde entonces, la competencia ha cambiado su enfoque hacia otros desafíos emergentes, como predecir la estructura de múltiples proteínas que interactúan en un complejo.
Las herramientas de IA están diseñando proteínas completamente nuevas que podrían transformar la medicina
Ahora, muchos esperan que estas competiciones puedan hacer avanzar el campo del diseño de proteínas, tal como CASP ayudó a impulsar una revolución en la predicción de la estructura de las proteínas. “AlphaFold no existiría sin CASP”, afirma Rust. “Necesitamos estas competiciones para hacer bien el trabajo y motivar a la gente”.
En junio, Rost y varios colegas ganaron el Campeonato de Ingeniería de Proteínas organizado por Align to Innovate, una organización internacional de ciencia abierta sin fines de lucro. El evento incluyó dos partes. Primero, los participantes intentaron predecir las propiedades de diferentes variantes de enzimas. Los equipos con mejor desempeño en esta ronda rediseñaron la enzima que descompone el almidón, y los mejores diseños se identificaron mediante experimentos de laboratorio. El torneo de 2025 ya está en marcha.
La competencia Winter Protein Design Games, que anunció su ganador en abril, fue dirigida por Liberum Bio, una empresa de biotecnología en Kitchener, Canadá, y Rosetta Commons, una colaboración de científicos en su mayoría académicos que mantienen herramientas de modelado de proteínas. La competencia encargó a los participantes la tarea de rediseñar una proteína existente (una enzima de virus vegetal ampliamente utilizada en la purificación de proteínas) para hacer que la molécula fuera más eficiente.
En otros dos concursos se pidió a los participantes que idearan proteínas completamente nuevas. Adaptyv buscaba proteínas capaces de unirse a un receptor de la hormona del crecimiento llamado EGFR que es hiperactivo en muchos tipos de cáncer. Los 90 participantes presentaron más de 700 diseños.
En el proyecto Bits to Binders, los investigadores compiten para producir pequeñas proteínas que puedan… Puede utilizarse en el tratamiento del cáncer de células T.. Está dirigido por la BioML Society, un grupo dirigido por estudiantes de posgrado de la Universidad de Texas en Austin, y ha atraído a 64 equipos de 42 países, incluidos Nigeria, Colombia, Irán e India. Actualmente se están probando unos 18.000 diseños y los resultados están previstos para principios de 2025. “Nos sorprendió mucho la participación”, dice el coorganizador Clayton Kusunoki, estudiante de doctorado en bioquímica de la universidad.
Bienvenidos los recién llegados
Muchos de los participantes del concurso trabajan en el campo de la ingeniería y el diseño de proteínas, afirma Julian Englert, director ejecutivo y cofundador de Adaptyv. Sin embargo, el concurso también recibió propuestas prometedoras de personas sin experiencia profesional en biología. Un participante de Irán hizo sus predicciones utilizando una computadora de juego porque no tenía acceso a sistemas más potentes.
Englert dice que las entradas de alta calidad de conocidos no investigadores le recuerdan los orígenes de las reparaciones en talleres de Apple, Microsoft y otros gigantes tecnológicos. “Les habría llevado dos años de estudio y unirse a un laboratorio para llegar al punto en el que pudieran empezar a hacerlo en un fin de semana”. Imagina un futuro en el que los diseñadores de proteínas independientes compitan por premios ofrecidos por las empresas. laboratorios académicos y otros investigadores de una molécula.
“Todo el mundo de las proteínas”: la IA predice la forma de casi todas las proteínas conocidas
Los concursos también pueden ahorrar tiempo de otras maneras. Obtener resultados experimentales rápidos de los organizadores del concurso fue un gran incentivo para Michael Heinsinger, científico de aprendizaje automático de la Universidad Técnica de Munich, que formó parte del equipo ganador con Rost. “De lo contrario, habríamos tenido que tomarnos el tiempo para redactar una subvención”, afirma. “Para mí, el premio es ahorrar tiempo”.
En términos de premios reales, el torneo Align to Innovate no ofrece ninguno, pero algunos sí. Los ganadores del concurso Bits to Binders recibirán una taza impresa en 3D con su diseño y algunos productos de la empresa de biotecnología que realiza las pruebas, llamada LEAH Laboratories en Eagan, Minnesota. También habrá oportunidades de colaboración.
Adaptyv, que vende pruebas de laboratorio automatizadas para moléculas creadas por diseñadores de proteínas, ha ofrecido varias pruebas gratuitas y algunos de sus propios productos. Los ganadores de los Juegos de Invierno de Rosetta recibieron 5.000 dólares.
Pero lo más destacado es la competencia Evolved 2024 recientemente lanzada, donde el equipo que obtenga el primer lugar recibirá un crédito de Amazon Web Services de $25,000, junto con créditos de otras compañías (incluida OpenAI) por valor de miles de dólares. Entre sus patrocinadores se incluyen Lux Capital, una firma de capital de riesgo de la ciudad de Nueva York que ha invertido más de 1.500 millones de dólares en empresas de tecnología, y Escala evolutivauna startup de inteligencia artificial biológica también en la ciudad de Nueva York, ha atraído una inversión de 142 millones de dólares.
Más allá de los grandes premios en efectivo
Elegir quién cosechará estas recompensas no siempre es fácil. La competencia Evolved 2024, que es similar a un hackathon, donde los equipos trabajan en problemas a gran escala, como predecir la eficacia y seguridad de los medicamentos, será juzgada en persona por un panel de expertos. Pero incluso en competencias con objetivos de diseño de proteínas más claros, “no es fácil saber quién va a ganar”, dice Erika DeBenedictis, ingeniera biológica y fundadora de Align to Innovate. El campeonato de su organización midió los diseños en función de su actividad, estabilidad y qué tan bien (o incluso posibles) podrían realizarse. “Cuando se diseña una proteína, hay muchas maneras en que puede fallar”, dice.
Para que las competencias transformen el diseño de proteínas, deberán abordar los desafíos que enfrenta este campo más amplio, dicen los científicos. A diferencia de la predicción de estructuras, el diseño de proteínas puede tener parámetros muy diferentes de una tarea a otra. Es posible que los métodos utilizados para formular un tipo particular de enzima no se traduzcan en otras proteínas, como los componentes de las vacunas.

¿Qué sigue para AlphaFold y la revolución del plegado de proteínas con IA?
Rost advierte que los concursos pueden resultar contraproducentes si empeoran la situación, por ejemplo, al juzgar los diseños de forma demasiado estricta. Es posible que los investigadores tampoco aprovechen todos los beneficios de los concursos de diseño de proteínas Si los concursantes guardan silencio sobre sus métodosdice Anthony Jeter, biólogo computacional de la Universidad de Wisconsin-Madison. “Si los equipos no comunican sus métodos, hay pocas oportunidades de aprender qué funciona y qué no”.
Hasta el momento, esto no parece estar sucediendo. La mayoría de los concursos alientan a los participantes o incluso les piden que describan sus métodos. Jeter dice que las competencias también pueden ayudar a reunir algunos de los campos dispares que se han sentido atraídos por el diseño de proteínas, desde laboratorios de bioquímica que han sido pioneros en métodos de ingeniería de proteínas, hasta científicos de aprendizaje automático que han sido pioneros en sus habilidades en el procesamiento del lenguaje natural. “Las personas que organizan competiciones, para tener el máximo impacto en el campo, deben pensar detenidamente cómo crear una comunidad”.
Cuando se anunciaron los resultados del concurso Adaptyv a finales de septiembre, Naka se sintió decepcionado. Aunque sus 10 entradas parecían sólidas, ninguno de sus diseños funcionó en el laboratorio. Sólo 5 de 147 diseños probados se unieron realmente a la molécula objetivo. No fue posible hacer más de 50 de ellos.
En realidad, esto no está mal: los esfuerzos anteriores para diseñar aglutinantes EGFR han tenido tasas de éxito mucho más bajas. “Es normal en el curso de ingeniería de proteínas: hay que estar preparado para fracasar muchas veces”, dice Naka. Los ganadores son Martin Pasisa y Lennart Nickel, biólogos estructurales del Instituto Federal Suizo de Tecnología (EPFL) en Lausana, quienes publicaron una preimpresión que describe su enfoque y convirtieron su código en código abierto (METRO. base et al. Preimpresión en bioRxiv https://doi.org/nmfm; 2024). Adaptyv lo tiene ahora Se lanzó el segundo concurso. Lo cual se basa en el primero.
Naka desearía haber empezado a trabajar en sus entradas antes. Describe su hackathon como “algo divertido”: doloroso en ese momento, pero divertido en retrospectiva. A través de la competencia, pudo entablar relaciones con científicos de ideas afines, incluido Jeter. “Es como si redujera la barrera de entrada y permitiera que muchas personas nuevas se involucraran en el diseño de proteínas”, dice. “Definitivamente participaré en eventos similares en el futuro”.