[ad_1]
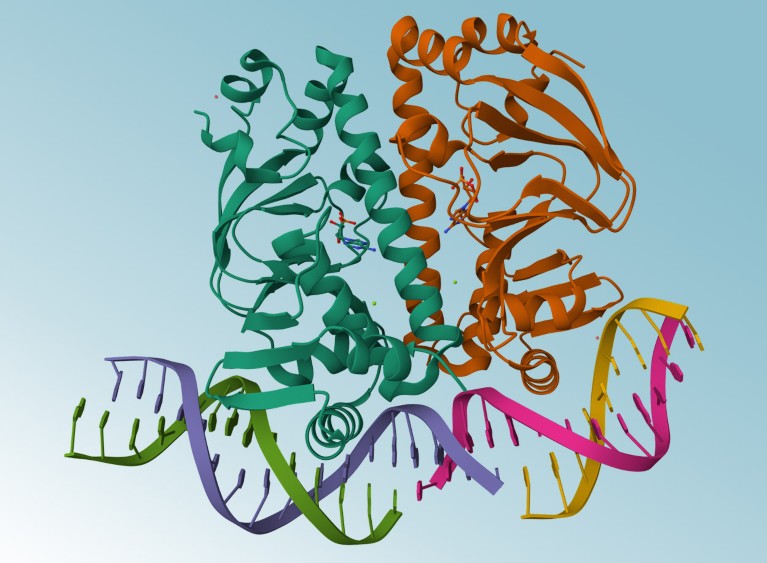
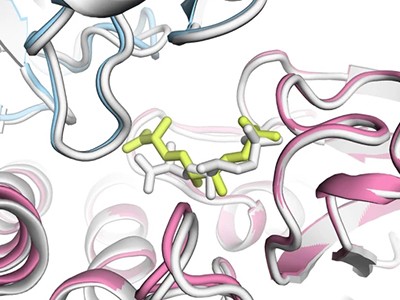
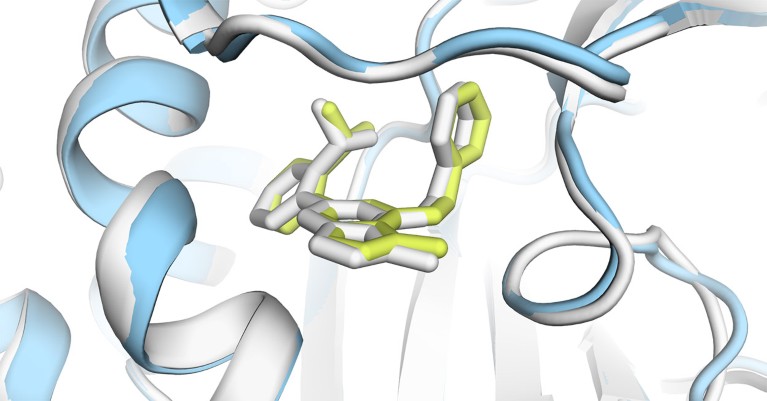
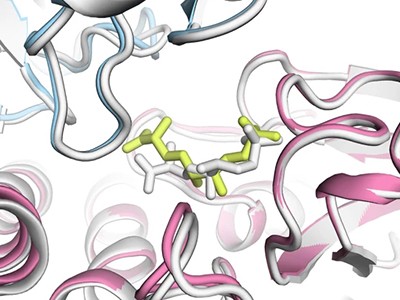
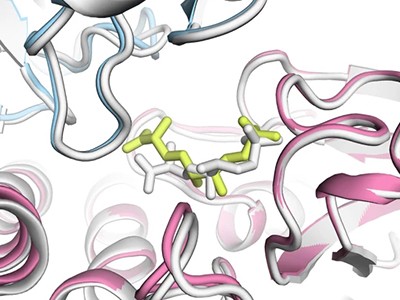
AlphaFold3 puede predecir la estructura de las proteínas cuando interactúan con el ADN.Crédito: Ferrell et al./Sociedad Estadounidense de Microbiología,mol*, RCSB PDB
AlphaFold3 finalmente está abierto. Seis meses después de Google DeepMind El código fue bloqueado polémicamente De un Un artículo que describe un modelo de predicción de la estructura de las proteínas.Los científicos ahora pueden Descarga el código del programa Y el uso de la herramienta de inteligencia artificial (IA) para aplicaciones no comerciales, como anunció el 11 de noviembre la compañía, con sede en Londres.
“Estamos muy emocionados de ver qué hace la gente con esto”, dijo el mes pasado John Jumper, quien dirige el equipo AlphaFold de DeepMind, junto con el director ejecutivo Demis Hassabis. Ganó una parte del Premio Nobel de Química 2024 Por su trabajo en la herramienta de inteligencia artificial.
La importante actualización de AlphaFold impulsa el descubrimiento de fármacos
AlphaFold3, a diferencia de sus predecesores, es Capaz de modelar proteínas en coordinación con otras moléculas.. Pero en lugar de publicar su código central, como sucedió con Pliegue alfa 2 DeepMind proporcionó acceso a través de un servidor web, restringiendo la cantidad y los tipos de predicciones que los científicos podían hacer.
Lo más importante es que el servidor AlphaFold3 impidió que los científicos predijeran cómo se comportarían las proteínas en presencia de posibles fármacos. Pero ahora, la decisión de DeepMind de publicar el código significa que los científicos académicos pueden predecir tales interacciones ejecutando el modelo ellos mismos.
Inicialmente, la compañía dijo que hacer que AlphaFold3 esté disponible solo a través de un servidor web logra el equilibrio adecuado entre permitir el acceso para la investigación y proteger las ambiciones comerciales. Isomorphic Labs, una filial londinense de DeepMind, está aplicando AlphaFold3 al descubrimiento de fármacos.
Pero implementar AlphaFold3 sin su código o pesos de modelo (parámetros obtenidos entrenando el programa en estructuras de proteínas y otros datos) Provocó críticas de los académicos.quien dijo que la medida socavaba el potencial reproductivo. DeepMind rápidamente cambió de rumbo y dijo que proporcionaría una versión de código abierto de la herramienta dentro de medio año.
Ahora cualquiera puede descargar el código del software AlphaFold3 y utilizarlo de forma no comercial. Pero actualmente, sólo los académicos con afiliación académica pueden acceder a las pesas de entrenamiento previa solicitud.
Versiones accesibles
DeepMind se ha enfrentado a la competencia: en los últimos meses, varias empresas se han enfrentado a ella Se presentan herramientas de predicción de estructuras de proteínas de código abierto basadas en AlphaFold3basado en las especificaciones descritas en el artículo original conocido como pseudocódigo.
Dos empresas chinas, el gigante tecnológico Baidu y el desarrollador de TikTok ByteDance, han lanzado sus propios modelos inspirados en Alpha Fold 3, al igual que una startup en San Francisco, California, llamada Zhai Discovery.
AlphaFold3 – ¿Por qué Nature lo publicó sin su código?
Una limitación importante de estos modelos es que, al igual que AlphaFold3, ninguno de ellos tiene licencia para aplicaciones comerciales, como el descubrimiento de fármacos, afirma Mohammed Al-Quraishi, biólogo computacional de la Universidad de Columbia en la ciudad de Nueva York. Sin embargo, el prototipo de Chai Discovery, Chai-1, podría utilizarse a través de un servidor web para este tipo de trabajo, afirma Jack Dent, uno de los fundadores de la empresa.
Otra empresa, Ligo Biosciences, con sede en San Francisco, ha lanzado una versión sin conexión de AlphaFold3. Pero todavía no tiene toda la gama de capacidades, incluida la capacidad de modelar fármacos y moléculas distintas de las proteínas.
Otros equipos están trabajando en versiones de AlphaFold3 que no tengan tales límites: Al-Quraishi espera tener disponible un modelo de código completamente abierto llamado OpenFold3 para fin de año. Esto permitiría a las empresas farmacéuticas volver a entrenar sus propias versiones del modelo utilizando datos patentados, como las estructuras de las proteínas unidas a diferentes fármacos, lo que podría conducir a un mejor rendimiento.
La apertura es importante
El año pasado se produjo una avalancha de nuevos modelos biológicos de IA lanzados por empresas con diferentes enfoques de apertura. Anthony Jeter, biólogo computacional de la Universidad de Wisconsin-Madison, no tiene ningún problema con que empresas con fines de lucro se unan a su campo, siempre y cuando sigan las mismas reglas que otros científicos cuando comparten su trabajo en revistas y servidores de preimpresión.
Jeter añade que si DeepMind hace afirmaciones sobre AlphaFold3 en una publicación científica, “yo y otros también esperaríamos que compartieran información sobre cómo hace predicciones y diseñaran sus modelos y códigos de IA de una manera que podamos examinar”. “Mi grupo no se basará en herramientas que no podamos examinar y utilizar”.
No todos los modelos de IA de “código abierto” son realmente abiertos: aquí hay un desglose
El hecho de que ya hayan surgido tantas copias de AlphaFold3 muestra que el modelo era replicable, incluso sin código fuente abierto, dice Pushmeet Kohli, jefe de IA para ciencia en DeepMind. Añade que en el futuro le gustaría ver más debates sobre los estándares de publicación en un campo donde el número de investigadores académicos y empresariales está aumentando.
La naturaleza de código abierto de AlphaFold2 ha dado lugar a una avalancha de innovaciones por parte de otros científicos. Por ejemplo, los ganadores de un reciente concurso de diseño de proteínas utilizaron una herramienta de inteligencia artificial para el diseño. Nuevas proteínas capaces de unirse al objetivo del cáncer. El reciente hack AlphaFold2 favorito de Jumper fue el de un equipo que usó la herramienta para Identificar la proteína clave que ayuda a los espermatozoides a unirse a los óvulos.
Jumper no puede esperar a que lleguen tales sorpresas después de la participación de AlphaFold3, incluso si no siempre se materializan. “La gente lo utilizará de formas extrañas”, predice. “A veces fracasarás y otras veces triunfarás”.
[ad_2]
Source Article Link