[ad_1]

Una técnica de seguimiento molecular conocida como PAPA-fSMT utiliza etiquetas fluorescentes para estudiar complejos de proteínas en la célula.Derechos de autor: Getty
Las células vivas están llenas de proteínas. Pero estas proteínas rara vez funcionan solas; Impulsan el comportamiento celular al emparejarse con otras proteínas para formar complejos temporales o duraderos. “La mayoría de las proteínas de la célula no son monógamas, por así decirlo”, dice Thomas Graham, biofísico de la Universidad de California, Berkeley.
Estas relaciones afectan la función: una proteína puede realizar muchas tareas celulares, dependiendo de las moléculas con las que interactúa.
Pero estudiar complejos proteicos en las células ha sido difícil. La mayoría de las técnicas requieren romper células para obtener proteínas para estudiar en un tubo de ensayo, o moléculas para marcar, pero a menudo las etiquetas están demasiado dispersas para que las interacciones se puedan observar de manera confiable. En 2022, Graham y sus colegas del laboratorio del bioquímico Robert Tjian de la Universidad de California, Berkeley, describieron una forma de capturar estas interacciones en células vivas a nivel de una sola molécula.1Este método, llamado fotoactivación asistida por proximidad (PAPA), utiliza la excitación de un fluoróforo con luz verde para reactivar un segundo fluoróforo cercano desde un estado oscuro. La mayoría de las proteínas que emiten fluorescencia están cerca unas de otras y, por lo tanto, es probable que formen el mismo complejo con la proteína que lleva el fluoróforo excitado. “El método PAPA nos permite, por primera vez, iluminar moléculas que interactúan con un compañero particular y observar esas interacciones por pares dentro de la célula”, dice Graham.
Nuevo y mejorado
En una preimpresión publicada el 25 de junio, el equipo se basa en esta técnica combinándola con el seguimiento rápido de una sola molécula fSMT para determinar la dinámica molecular de un paso clave en la regulación de la transcripción genética.2. Llaman al nuevo método PAPA-fSMT.
“El trabajo anterior fue una muy buena prueba de concepto, pero aquí dieron un paso más al agregar el seguimiento de una sola molécula para poder medir la difusión del complejo proteico así como la proteína individual. “Han demostrado por primera vez esta cinética en células vivas”.
Graham dice que PAPA fue descubierto por casualidad durante el cierre de Covid-19. Como parte del estudio del nucléolo, una estructura del núcleo donde se ensamblan los ribosomas, Graham ha estado utilizando longitudes de onda específicas para alternar entre la excitación de un tinte rojo fluorescente y uno verde. Como era de esperar, las moléculas rojas desaparecieron rápidamente debido al fotoblanqueo, cambiando su estructura molecular para que no pudieran ser fluorescentes. Pero a medida que continuaba cambiando de longitud de onda, el pigmento rojo apareció de nuevo: algunas de las moléculas entraron en un estado oscuro sin ser fotoblanqueadas y aparentemente se activaron indirectamente mediante la excitación de la luz verde (ver “Interacciones informativas”). Estudios adicionales revelaron que para reactivar la proteína marcada en rojo, debe estar cerca de la proteína marcada en verde, lo que indica que es probable que las dos proteínas estén unidas en el mismo complejo. “Fue muy inesperado. Nunca antes había visto algo así”, dice Graham.
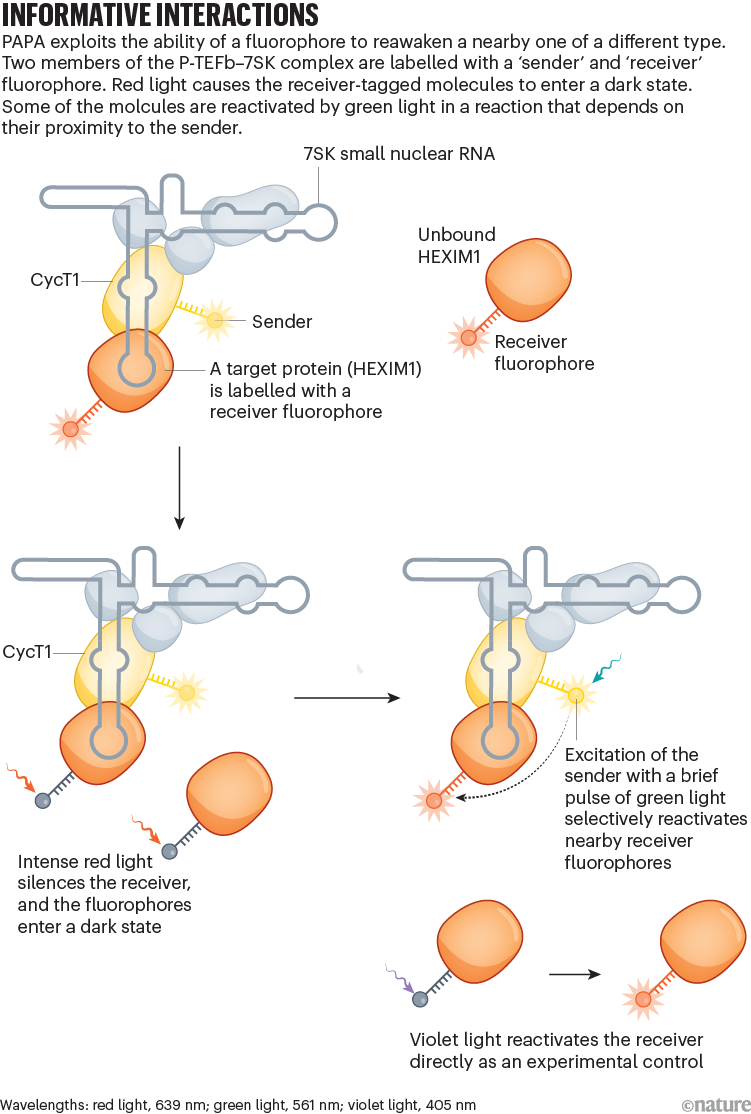
La señal de reactivación era más fuerte cuando las moléculas estaban muy cerca (sólo 68 angstroms, o aproximadamente 3,5 veces el diámetro de la doble hélice del ADN), pero seguía siendo detectable a una distancia de más de 2,5 veces esa distancia.
Aunque Graham aún no ha podido identificar la gama completa de tintes compatibles, ha observado el efecto utilizando tintes de rodamina y carborhodamina, así como algunas proteínas fluorescentes. Todavía no está claro exactamente cómo funciona la reactivación, pero la técnica ofrece una forma valiosa de investigar selectivamente compuestos específicos en células vivas, dice Taekjib Ha, biofísico de la Facultad de Medicina de Harvard.
Ha se especializa en otro enfoque para estudiar la proximidad molecular en las células: la transferencia de energía por resonancia de fluorescencia (FRET), en la que un fluoróforo transfiere su energía de excitación a un fluoróforo cercano. La transferencia de energía puede explicar por qué PAPA también funciona, pero las técnicas funcionan a diferentes distancias: la señal FRET decae aproximadamente a la mitad de la distancia que decae la señal PAPA. Además, en FRET, las proteínas están marcadas tan escasamente que la posibilidad de unir dos compuestos es mucho menor.
Graham señala que, a diferencia de la resonancia magnética fluorescente, los dos tintes en PAPA se excitan en momentos diferentes, por lo que no hay interferencia de la fluorescencia de fondo. Esto es importante para obtener imágenes de una sola molécula en altas concentraciones de proteínas.
Dinámica molecular
En la preimpresión del artículo, Graham y sus colegas exploraron cómo la quinasa P-TEFb activa la transcripción. Normalmente, cuando la ARN polimerasa comienza a transcribir un gen, detiene el proceso después de copiar las primeras docenas de bases; P-TEFb restaura la transcripción, pero cuando se une al complejo de ribonucleoproteína 7SK, esta actividad se inhibe. Los investigadores han nombrado una subunidad de la enzima P-TEFb, así como HEXIM1, una subunidad del complejo 7SK. El equipo aplicó PAPA a cuatro pares de proteínas y descubrió que el complejo P-TEFb-7SK se difunde más lentamente, de acuerdo con su mayor peso molecular, pero en su mayor parte no está asociado con la cromatina, lo que sugiere que el complejo generalmente existe sin unirse al ADN. La adición de un inhibidor de P-TEFb al sistema disoció rápidamente P-TEFb y HEXIM1. “Vemos que todo el complejo colapsa simultáneamente cuando las células se tratan con este inhibidor”, dice Graham.
En su propia investigación sobre la separación de fases, el fenómeno en el que las moléculas dentro de las células se separan espontáneamente en compartimentos celulares esencialmente distintos, Myung propone varias estrategias para combinar PAPA con el seguimiento de moléculas individuales. Myung señala que esta técnica no puede revelar nuevos actores en los complejos que estudia, porque los investigadores deben conocer las identidades de los componentes para etiquetarlos. “Pero si tienes las proteínas correctas etiquetadas, puedes intentar mapear estas cosas una por una, lo cual es realmente genial”.
Mientras tanto, Graham y sus colegas continúan trabajando en PAPA para completar los detalles moleculares de la transcripción. Actualmente, Graham utiliza esta técnica para determinar si las moléculas interactúan o no, pero también para determinar cuánto tiempo continúan interactuando; También pretende mejorar la especificidad de esta técnica mediante el desarrollo de métodos que tengan en cuenta la reactivación no específica, que aparece como ruido de fondo que puede distorsionar la imagen.
Los nuevos dispositivos también pueden ayudar. Graham está colaborando con los biofísicos Eric Betzig y Sreejokul Upadhyayula y su investigador postdoctoral Amir Hai en UC Berkeley para construir un nuevo tipo de microscopio que pueda capturar una mayor cantidad de trayectorias de moléculas individuales. “Existe todo un universo de interacciones entre proteínas que se pueden explorar en las células vivas”, afirma Graham. Ahora, gracias a PAPA-fSMT, los investigadores finalmente podrán intentar estudiarlo donde existe, en lugar de en tubos de ensayo.
[ad_2]
Source Article Link

