[ad_1]
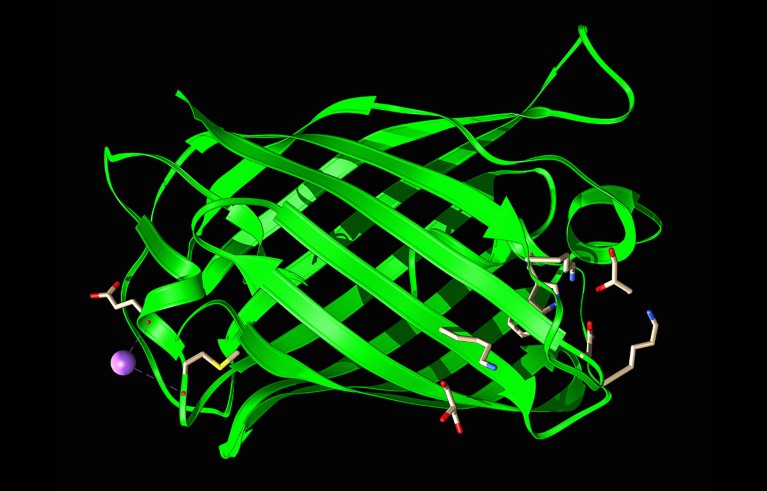
Modelo estructural de proteína verde fluorescente, elemento fundamental en biotecnología.Copyright: Biblioteca de imágenes científicas/Laguna Design
a inteligencia artificial (Inteligencia Artificial) modelo parlante El lenguaje de las proteínas. – Una de las mayores aplicaciones desarrolladas hasta la fecha para la biología: se ha utilizado para crear nuevas moléculas fluorescentes.
La oferta de prueba de principio fue anunciada este mes por EvolutionaryScale, con sede en la ciudad de Nueva York, junto con $142 millones en nuevos fondos para aplicar su modelo a… Desarrollo de fármacosLa empresa, fundada por científicos que anteriormente trabajaron en el gigante tecnológico Meta, se especializa en aplicar modelos de aprendizaje automático de vanguardia entrenados en lenguaje e imágenes a datos biológicos.
Las herramientas de IA están diseñando proteínas completamente nuevas que podrían transformar la medicina
“Queremos crear herramientas que puedan hacer que la biología sea programable”, dice Alex Reeves, científico jefe de la compañía, quien ha sido parte del esfuerzo de Meta para aplicar inteligencia artificial a datos biológicos.
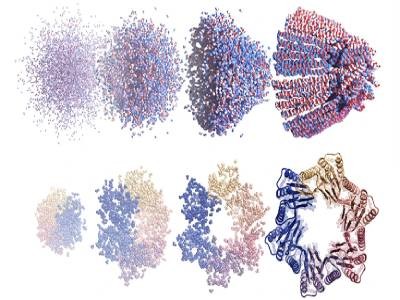
La herramienta de inteligencia artificial de EvolutionaryScale, llamada ESM3, es lo que se conoce como modelo de lenguaje de proteínas. Fue entrenado en más de 2.700 millones de secuencias y estructuras de proteínas, así como en información sobre las funciones de estas proteínas. El formulario se puede utilizar Creando proteínas Para especificaciones proporcionadas por los usuarios, similar al texto generado por chatbots como ChatGPT.
“Este será uno de los modelos de IA en biología al que todo el mundo prestará atención”, afirma Anthony Jeter, biólogo computacional de la Universidad de Wisconsin-Madison.
brillante
Reeves y sus colegas habían trabajado en iteraciones anteriores del modelo ESM en Meta, pero comenzaron a trabajar por su cuenta el año pasado, después de que Meta terminara su trabajo en esta área. Anteriormente utilizaron el modelo ESM-2 para crear el modelo ESM-2. Una base de datos de libre acceso que contiene 600 millones de estructuras proteicas predichas1Desde entonces, otros equipos han utilizado versiones de ESM-1 para diseñar anticuerpos con actividad mejorada contra patógenos, incluido el SARS-CoV-2.2 y rediseñar proteínas “anti-CRISPR” para mejorar la eficiencia de las herramientas de edición de genes3.
Este año, otra empresa especializada en IA en biología, Profluent en Berkeley, California, utilizó su modelo de lenguaje de proteínas para crear nuevas proteínas de edición de genes inspiradas en CRISPR, y puso una de estas moléculas a disposición de su uso de forma gratuita.
Para probar su último modelo, el equipo de Reeves se propuso probar otra herramienta biotecnológica: la proteína verde fluorescente (GFP), que absorbe la luz azul y brilla en verde. Los investigadores aislaron la proteína verde fluorescente en la década de 1960 a partir de medusas bioluminiscentes. Igual victoriaInvestigaciones posteriores, que recibieron el Premio Nobel con este descubrimiento, mostraron cómo la proteína verde fluorescente podía marcar otras proteínas cuando se observaban al microscopio, explicaron las bases moleculares de la fluorescencia de las proteínas y desarrollaron versiones artificiales de la proteína que brillan más intensamente y en Colores diferentes.
Desde entonces, los investigadores han identificado otras proteínas fluorescentes con una forma similar, todas las cuales comparten un núcleo de “pigmento” emisor y absorbente de luz rodeado por un andamio en forma de barril. El equipo de Reeves pidió a ESM3 que creara ejemplos de proteínas similares a proteínas fluorescentes verdes que contengan un conjunto de aminoácidos clave que se encuentran en el cromóforo de la proteína fluorescente verde.
Los investigadores sintetizaron 88 de los diseños más prometedores y midieron su capacidad de fluorescencia. La mayoría no tuvo éxito, pero un diseño, diferente de las proteínas fluorescentes conocidas, brillaba débilmente, unas 50 veces más débil que las formas naturales de GFP. Utilizando la secuencia de esta molécula como punto de partida, los investigadores encargaron al ESM3 la tarea de mejorar su trabajo. Cuando los investigadores hicieron alrededor de 100 de los diseños resultantes, muchos eran tan brillantes como la GFP natural, que sigue siendo mucho más débil que las variantes diseñadas en laboratorio.
Se prevé que una de las proteínas más brillantes diseñadas por ESM3, llamada esmGFP, tenga una estructura similar a la de las proteínas fluorescentes naturales. Sin embargo, su secuencia de aminoácidos es muy diferente y coincide con menos del 60% de las secuencias de proteínas fluorescentes más estrechamente relacionadas en su conjunto de datos de entrenamiento. Se publicó una preimpresión en el servidor bioRxiv.4Reeves y sus colegas dicen que, basándose en las tasas de mutación naturales, este nivel de variación de secuencia equivale a “más de 500 millones de años de evolución”.
Pero Jeter teme que esta comparación sea una forma inútil y quizás engañosa de describir el producto de un modelo de IA de vanguardia. “Suena aterrador cuando se piensa en la IA y en la aceleración del desarrollo”, afirma. “Siento que sobreestimar lo que hace un modelo puede ser perjudicial para el campo y potencialmente peligroso para el público”.
Reeves sostiene que la generación de nuevas proteínas mediante la duplicación de diferentes secuencias por parte de ESM3 es similar a la evolución. “Creemos que es interesante la perspectiva que necesitaría la naturaleza para poder generar algo como esto”, añade.
Umbral de riesgo
El ESM-3 se encuentra entre los primeros modelos de IA biológica que utiliza suficiente potencia informática durante su entrenamiento para obligar a los desarrolladores a notificar e informar al gobierno de EE. UU. Medidas de mitigación de riesgosSegún una orden ejecutiva presidencial emitida en 2023, EvolutionaryScale dice que ya ha estado en contacto con la Oficina de Política Científica y Tecnológica de EE. UU.
¿Podrían utilizarse como arma las proteínas diseñadas por IA? Los científicos determinan pautas de seguridad
La versión de ESM3 que supera este umbral, que consta de alrededor de 100 mil millones de parámetros o variables que el modelo utiliza para representar relaciones entre secuencias, no está disponible públicamente. Para una versión más pequeña y de código abierto, se excluyeron del entrenamiento ciertas secuencias, como las que pertenecen a virus y la lista de patógenos y toxinas preocupantes del gobierno de EE. UU. ESM3-open, que los científicos pueden descargar y ejecutar de forma independiente en cualquier lugar, tampoco puede generar tales proteínas.
Martin Pachisa, biólogo estructural del Instituto Federal Suizo de Tecnología en Lausana, está entusiasmado de empezar a trabajar con ESM3. Señala que es uno de los primeros modelos biológicos que permite a los investigadores especificar diseños utilizando descripciones en lenguaje natural de sus propiedades y funciones, y está interesado en ver cómo funcionan experimentalmente estas y otras características.
A Basisa le impresionó que EvolutionaryScale lanzara una versión de código abierto de ESM3 y una descripción clara de cómo entrenar la versión más grande. Pero un modelo más grande requeriría vastos recursos informáticos para desarrollarse de forma independiente, afirmó. “Ningún laboratorio académico podrá replicarlo”.
Reeves espera aplicar ESM-3 a otros diseños. Será interesante ver cómo resulta ESM-3, dice Pachesa, quien formó parte del equipo que utilizó un paradigma de lenguaje de proteínas diferente para crear nuevas proteínas CRISPR. Reeves imagina aplicaciones en sostenibilidad (un vídeo en su sitio web muestra el diseño de enzimas que comen plástico) y en el desarrollo de anticuerpos y otros medicamentos basados en proteínas. “Es realmente un modelo a la vanguardia”, afirma.
[ad_2]
Source Article Link