[ad_1]

Un grupo tecnológico en Cambridge, Massachusetts, permite a investigadores de diferentes sectores colaborar fácilmente.Crédito: Brooks Craft LLC/Corbis/Getty
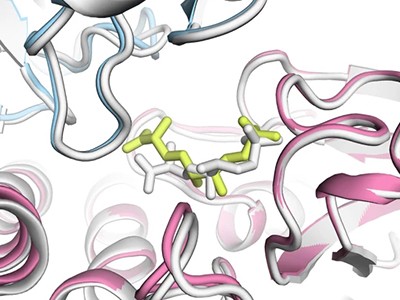
La última versión del algoritmo de predicción de la estructura de proteínas AlphaFold ha generado un gran interés desde su lanzamiento, acompañada de un artículo en naturalezaa principios de este mes1. A diferencia de su predecesor AlphaFold2, AlphaFold3 puede predecir no sólo las estructuras de los complejos de proteínas, sino también cuándo las proteínas interactúan con otros tipos de moléculas, incluidos el ADN y el ARN. La herramienta de IA será importante tanto en la investigación básica como en el descubrimiento de fármacos.
Pero su lanzamiento también generó preguntas y críticas tanto del equipo AlphaFold de Google DeepMind en Londres como… naturaleza. Cuando se publicó AlphaFold22El código central completo ahora es accesible para todos los investigadores. Pero AlphaFold3 viene con un “pseudocódigo”: una descripción detallada de lo que el código puede hacer y cómo funciona.
La importante actualización de AlphaFold impulsa el descubrimiento de fármacos
Esta no fue una decisión que tomamos a la ligera y este editorial explica sucintamente nuestras razones. Creemos que la investigación, independientemente del sector en el que se realice, debe evaluarse mediante revisión por pares y publicarse en beneficio de la sociedad y la ciencia. Al mismo tiempo, no queremos que ésta sea la última palabra. Esta es una oportunidad para mantener una conversación importante entre todas las partes interesadas en la investigación en un momento en que la mayoría de la investigación mundial recibe financiación privada.
Los conceptos básicos de cómo la comunidad utilizará la nueva versión de AlphaFold siguen siendo los mismos: cualquier persona con una cuenta de Google puede utilizar la herramienta de forma gratuita para aplicaciones no comerciales. Pero existen diferencias importantes entre la última versión y las versiones anteriores. Vale la pena señalar que para AlphaFold2, el equipo de DeepMind trabajó con el Instituto Europeo de Bioinformática del Laboratorio Europeo de Biología Molecular, una organización de investigación intergubernamental (y financiada con fondos públicos) con sede en Cambridge, Reino Unido. Los investigadores de DeepMind entrenaron sus programas en miles de registros publicados de estructuras de proteínas y sus conformaciones conocidas.
Ahora, DeepMind se ha asociado con Isomorphic Labs, una empresa de desarrollo de fármacos con sede en Londres propiedad de Alphabet, la empresa matriz de Google. Además de la falta de disponibilidad del código completo, existen otras limitaciones en el uso de la herramienta, por ejemplo, en el desarrollo de fármacos. También existen límites diarios en la cantidad de predicciones que pueden hacer los investigadores individuales.
Muchos investigadores están decepcionados por estas limitaciones. En una carta abierta a naturaleza (ser visto go.nature.com/3k9acav), sus autores señalan que la falta de código fuente abierto limita los esfuerzos de verificación y replicación. El mensaje tuvo impacto. Poco después de su publicación, el vicepresidente de investigación de DeepMind, Pushmeet Kohli, publicó en la plataforma de redes sociales Éste es un paso importante y naturaleza La hoja publicada se actualizará una vez que se publique el código.
La capacidad de la IA para acelerar el descubrimiento de fármacos necesita una revisión de la realidad
Pero, ¿por qué permitimos que se restrinjan todos los datos y el código en el momento de la implementación? naturalezaLas políticas editoriales de se desarrollaron para apoyar la ciencia y el estado abiertos: “Se solicita a los autores que pongan los materiales, datos, códigos y protocolos asociados inmediatamente a disposición de los lectores sin calificaciones indebidas”. Una forma de capacitar a los investigadores para que hagan esto es asociándonos con Code Ocean, una plataforma de ciencia computacional que hace que la información sea repetible y rastreable. Pero nuestras políticas también reconocen la posibilidad de limitaciones, que “deben revelarse a los editores en el momento de la presentación” y acordarse con los editores. La política también establece: “Cualquier motivo que impida la necesidad de compartir código o algoritmo será evaluado por los editores, quienes se reservan el derecho de rechazar el artículo si el código crítico no está disponible”.
Las razones de las limitaciones incluyen la falta de estándares de presentación de datos a nivel disciplinario o la infraestructura tecnológica necesaria para depositar datos abiertamente y en repositorios estructurados. En otros casos, es posible que sea necesario proteger la confidencialidad o que los datos se retengan por motivos legales o de seguridad. Se aplican principios similares a la disponibilidad del código, y he aquí por qué naturalezaNuestras políticas incluyen la opción de publicar el modelo de entrenamiento y el pseudocódigo o código limitado, como lo demuestran los estudios de ejemplo que hemos publicado en Chemistry.3Cambio climático4 y virología5.
La mayoría de los proyectos de I+D en todo el mundo están financiados por el sector privado y muchos de los resultados de dichos trabajos no se publican en revistas revisadas por pares. estamos en naturaleza Creo que es importante que las revistas interactúen con el sector privado y trabajen con sus científicos para que puedan enviar sus investigaciones para revisión por pares y publicación. Esto mejora el intercambio de conocimientos, la validación de la investigación y la reproducibilidad que los investigadores se esfuerzan por lograr. También beneficia la seguridad y eficacia del producto. El progreso necesita más, no menos, datos abiertos, código, algo naturaleza Seguirá apoyando.
Pero este objetivo no se logrará en un solo paso. Requerirá una operación. Esto requiere participación y diálogo entre todas las partes interesadas. Repito: no queremos que nuestra opinión sea la última palabra, sino el inicio de la conversación. ¿Qué medidas adicionales podemos tomar para garantizar la apertura de todas las partes del ecosistema de investigación? Nos gustaría escuchar a todas las partes interesadas, no sólo a investigadores de universidades y empresas privadas, sino también a formuladores de políticas, ONG y editores. Escríbanos a [email protected]. Usaremos estas páginas para actualizar a los lectores.
[ad_2]
Source Article Link